



Por: Maria do Carmo de Freire Bastos
Extensas perdas econômicas associadas à agricultura são derivadas de doenças causadas por microrganismos em plantas de importância agronômica. Essas doenças têm como importantes agentes bactérias e fungos fitopatogênicos.
As medidas de controle dessas infecções geralmente envolvem o uso extensivo de pesticidas. Entretanto, tal estratégia apresenta diversas desvantagens, entre as quais vale a pena destacar os custos desse tratamento para o agricultor, a seleção de microrganismos resistentes aos pesticidas empregados e os riscos ambientais oriundos do acúmulo dessas substâncias no solo. Por isso, outra forma de se evitar tais infecções envolve a obtenção de plantas carreando genes naturais de resistência aos fitopatógenos. Entretanto, dependendo da planta, esta estratégia possui uso limitado devido à escassez desses genes de resistência naturais e ao longo período necessário para o cruzamento de plantas relacionadas, isto é, sexualmente compatíveis. Assim sendo, outra possibilidade hodierna que se tem mostrado bastante promissora é a obtenção de plantas carreando genes codificadores de peptídeos antimicrobianos de amplo espectro de ação (PAMs).
Os PAMs são produzidos por uma variedade de organismos procarióticos e eucarióticos, incluindo-se microrganismos, plantas, animais invertebrados e vertebrados (Quadro 1). Eles são considerados parte integrante da imunidade inata e a primeira linha de defesa das células contra microrganismos invasores (Jenssen et al., 2006). A maioria desses PAMs é catiônica e hidrofóbica a age ao nível da membrana citoplasmática das células-alvo, levando-as à morte. Entretanto, alguns desses PAMs possuem outros sítios-alvo de ação, como a parede celular, hidrolisando-a.
PAMs de diversas origens têm sido utilizados para a obtenção de plantas transgênicas. As plantas transgênicas são definidas como aquelas plantas carreando novos genes, oriundos de outras plantas, relacionadas ou não, de microrganismos ou de animais. A introdução desses genes nessas plantas é realizada por meio de experimentos de transformação, que, neste caso, representa a implantação de novos genes por mecanismos moleculares que não envolvem cruzamento e fecundação.
Quadro 1. Alguns exemplos de PAMs produzidos por organismos eucarióticos e procarióticos.
| Organismo Produtor | PAMs |
| Mamíferos | Defensivas, protegrina |
| Anfíbios | Magainina, bombinina, demaseptinas |
| Insetos | Defensinas, cecropinas, melitina |
| Plantas | Defensinas, tioninas, AP24 |
| Bactérias | Bacteriocinas |
As etapas para o desenvolvimento de uma planta transgênica incluem:
1. Seleção do gene de interesse;
2. Clonagem do gene em um vetor de expressão em eucarioto;
3. Transformação da planta escolhida com o vetor recombinante;
4. Integração do gene de interesse ao genoma da planta;
5. Seleção das plantas transformadas;
6. Expressão do gene de interesse e
7. Transmissão do gene aos descendentes.
Como exemplo, iremos abordar a obtenção de plantas transgênicas simples, duplas ou triplas de um cultivar de batata (Solanum tuberosum cv. Spunta), carreando os genes estruturais dos PAMs dermaseptina, lisozima e AP24, descrita no trabalho publicado por Rivero e colaboradores (2012). As características dos três PAMs estão sumarizadas no Quadro 2. Para a obtenção das plantas transgênicas, foi empregada a técnica de transformação baseada na utilização dos plasmídeos Ti (Kado, 1993).
Quadro 2. Características dos PAMs empregados para a obtenção de plantas transgênicas de Solanum tuberosum cv. Spunta.
| PAM | Fonte | Tamanho | Alvo de Ação | Microrganismos-alvo |
| dermaseptina | sapo | 28 a 34aa | membrana citoplasmática |
bactérias, fungos filamentosos, leveduras e protozoários |
| AP24 | Tabaco | cerca de 210aa | ||
| ovo de galinha | 129aa | parede celular | bactérias |
aa, ácidos aminados
Plasmídeos Ti
Os plasmídeos Ti (de indução de tumor) são megaplasmídeos de cerca de 200 kb encontrados em algumas estirpes de Agrobacterium tumefaciens, uma bactéria Gram-negativa encontrada no solo (Kado, 1993; Schröder & Lanka, 2005). Em sua organização genética, os plasmídeos Ti possuem duas regiões muito importantes para a formação de tumores nas plantas (Figura 1):
1. Região do DNA T. Esta região tem cerca de 25 kb e é ladeada por repetições diretas de 25 pb, chamadas bordos direito e esquerdo, e contém genes estruturais codificadores de auxinas e citocininas (ambas representando hormônios de crescimento vegetal) e de opinas (que são ácidos aminados condensados a açúcares), entre as quais predominam a octopina ou a nopalina. É esta região do DNA T que o A. tumefaciens é capaz de transferir para as plantas, gerando a formação de tumores.
2. Região vir. Esta região tem cerca de 35 kb e 25 genes vir (de virulência), agrupados em seis ou sete operons. Estes genes codificam todas as proteínas necessárias à formação de um canal intermembrana, entre a bactéria e a célula vegetal, e à transferência do DNA T para esta célula.
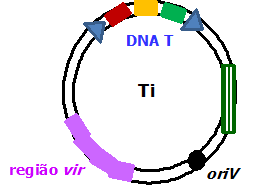
Figura 1. Esquema simplificado dos plasmídeos Ti, mostrando a região vir e a região do DNA T. Os triângulos azuis representam os bordos direito e esquerdo, os retângulos vermelho, amarelo e verde representam, respectivamente, os genes estruturais das auxinas, citocininas e opinas, e o retângulo verde riscado representa os genes envolvidos no catabolismo das opinas. Os vários plasmídeos Ti diferenciam-se, entre outros aspectos, pelo tipo de opina codificado e pelos genes responsáveis pelo seu catabolismo.
Quando o DNA T é transferido para a planta (somente uma das fitas do DNA é transferida), após contato celular estabelecido entre a bactéria e a célula vegetal (Figura 2), este DNA é transportado para o núcleo da célula, onde o DNA T se integra a um dos cromossomos, de uma forma randômica. Como os genes das auxinas, das citocininas e das opinas possuem promotores eucarióticos, após a integração, eles podem se expressar. A síntese das auxinas e das citocininas leva a uma multiplicação descontrolada das células vegetais, resultando na formação de tumores nas plantas. Já as opinas produzidas serão excretadas pelas células tumorais e servirão de fonte de C e de N para a nutrição bacteriana.
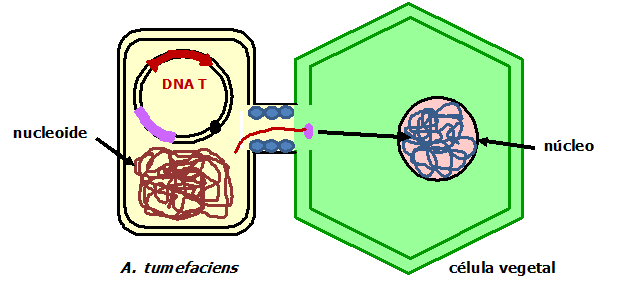
Figura 2. Esquema simplificado da transferência de uma fita do DNA T do plasmídeo Ti para a planta. A fita do DNA T é liberada do plasmídeo mediante a ação principal de duas proteínas Vir (VirD1 e VirD2), sendo que VirD2 () permanece covalentemente ligada ao extremo 5’ P da fita T liberada). Em seguida, a fita T é transferida para a célula vegetal mediante a formação de um canal intermembrana, ligando as duas células, formado por várias proteínas Vir (compondo um sistema de secreção do tipo 4). Uma vez no citoplasma da célula vegetal, a fita T é transferida para o núcleo, mediante a participação de proteínas Vir e de proteínas vegetais envolvidas em localização nuclear, e depois se integra a um cromossomo, passando a expressar os genes nela contidos.
Obtenção das plantas transgênicas contendo os genes estruturais de PAMs
Inicialmente, os genes codificadores da dermaseptina (De), da lisozima (Ly) e do AP24 (Ap) foram, separadamente, clonados em vetores de expressão apropriados. A clonagem foi feita dentro da região do DNA T, sendo que os genes dos hormônios de crescimento e da opina foram substituídos pelo gene de interesse, como mostrado na Figura 3. Os plasmídeos recombinantes gerados (pDe, pLy, pAp e pApLy) foram introduzidos no cultivar de batata escolhido (Spunta) por meio de transformação binária (Murai, 2013).
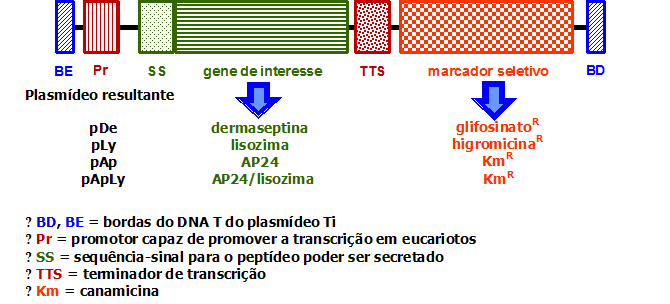
Figura 3. Características das sequências de DNA clonadas, carreando os genes estruturais codificadores de PAMs.
Na transformação binária, a planta é infectada por uma estirpe de A. tumefaciens carreando dois plasmídeos: um plasmídeo Ti, com deleção (D) de toda a região do DNA T, mas portador da região vir, e o plasmídeo recombinante contendo o gene de interesse e um gene marcador clonados na região do DNA T modificada, que só conserva os bordos direito e esquerdo. Os genes vir localizados no plasmídeo DTi irão promover a liberação da fita T, localizada no plasmídeo recombinante, e a sua transferência para as células vegetais. No caso da batata, foram empregados discos de minitubérculos para a infecção. Assim sendo, quando em contato com esses discos, a estirpe de A. tumefaciens pôde transferir o gene de interesse e o gene marcador para as células vegetais, mas não pôde mais causar a formação de tumores. Após a infecção, os minitubérculos foram, então, transferidos para um meio de cultivo adequado contendo o agente seletivo apropriado (glifosinato, higromicina ou canamicina). Após cerca de três semanas, foram obtidas plântulas resistentes aos agentes seletivos, que foram, então, crescidas em frascos com meio de cultura e, depois, transferidas para vasos com solo estéril, gerando as “explantas” (plantas derivadas de transformação).
A confirmação da presença dos genes de interesse nas “explantas” foi feita por PCR, empregando-se DNA extraído das folhas. A planta transgênica contendo o gene De foi, posteriormente, retransformada com o plasmídeo recombinante pApLy, gerando uma planta transgênica tripla, DeApLy, resistente ao glifosinato e à canamicina.
Os cinco tipos de plantas transgênicas (De, Ly, Ap, ApLy e DeApLy) foram testados quanto à resistência a cinco fitopatógenos de batateiras: duas espécies de bactérias (Erwinia caratovora eStreptomyces scabies) e três espécies de fungos (Phytophthora infestans, Rhizoctonia solani e Fusarium solani), empregando-se diferentes modelos de infecção: broto axial, folhas ou tubérculos. Os resultados obtidos estão sumarizados no Quadro 3.
++++, resistência >80%; +++, resistência entre 61 e 80%; ++, resistência entre 41 e 60%; +, resistência entre 21 e 40%; ±, resistência entre 5 e 20%; -, resistência <5%. Este nível de resistência foi determinado em função do número de plantas ou tubérculos testados (geralmente, em torno de 60 plantas ou tubérculos por tipo, oriundos de três experimentos independentes) que apresentaram sintomas e da gravidade dos sintomas observados: necrose, crestamento, clorose ou podridão.
Baseando-se nos experimentos realizados, pôde-se concluir:
● que as plantas transgênicas De, Ly, ApLy e DeApLy exibiram um alto nível de resistência aos patógenos bacterianos;
● que o nível de resistência aos fungos variou dependendo do gene presente na planta transgênica ou da combinação desses genes;
● que as plantas transgênicas duplas ou triplas foram as que exibiram um maior nível de resistência aos cinco patógenos testados, que são muito frequentes nas áreas de cultivo de batatas.
Os resultados aqui resumidos indicam que PAMs podem funcionar como uma nova fonte de resistência das plantas a um amplo grupo de fitopatógenos, podendo ser utilizados na prevenção de doenças de impacto econômico. Entretanto, embora os resultados apresentados neste trabalho sejam bastante promissores, demonstrando, de forma inequívoca, o potencial biotecnológico de PAMs também na agricultura, a efetividade dessas substâncias produzidas plantas transgênicas precisa ainda ser comprovada em experimentos de campo, em diferentes ecossistemas agrários, onde ocorre a interferência de fatores abióticos e bióticos, não levados em consideração nos experimentos realizados em condições ambientais controladas e em solo ou meio de cultivo estéril.
Referências:
Jenssen, H., Hamill, P. & and Hancock, R.E.W. 2006. Peptide antimicrobial agents. Clin. Microbiol. Rev., 19: 491-511.
Kado, C.I. 1993. Agrobacterium-mediated transfer and stable incorporation of foreign genes in plants. In: Bacterial Conjugation. Edited by D.B. Clewell, Plenum Press, New York, pp. 243-254.
Murai, N. 2013. Plant binary vectors of Ti plasmid in Agrobacterium tumefaciens with a broad host-range replicon of pRK2, pRi, pSa or pVS1. Am. J. Plant Sci., 4: 932-939.
Rivero, M., Furman, N., Mencacci, N., Picca, P., Toum, L., Lentz, E., Bravo-Almonacid, F. & Mentaberry, A. 2012. Stacking of antimicrobial genes in potato transgenic plants confers increased resistance to bacterial and fungal pathogens. J. Biotechnol., 157: 334-343.
Schröder, G. & Lanka, E. 2005. The mating pair formation system of conjugative plasmids - A versatile secretion machinery for transfer of proteins and DNA. Plasmid, 54: 1-25.
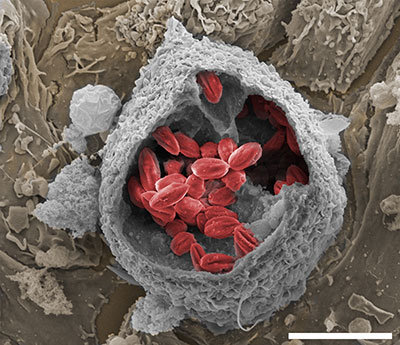 Imagem ampliada 7.500 vezes mostra as bolsas (vacúolos) formados pelo protozoário L. amazonensis (vermelho), agente causador da leishmaniose cutânea, dentro de macrófago de camundongo (cinza). FOTO Fernando Real/CNPQ
Imagem ampliada 7.500 vezes mostra as bolsas (vacúolos) formados pelo protozoário L. amazonensis (vermelho), agente causador da leishmaniose cutânea, dentro de macrófago de camundongo (cinza). FOTO Fernando Real/CNPQ
Pesquisadores da Escola Paulista de Medicina da Universidade Federal de São Paulo (Unifesp) identificaram novas pistas de como o parasita causador da leishmaniose invade as células de defesa do organismo e, uma vez no interior delas, retarda a ativação do arsenal imunológico que deveria eliminá-lo. O grupo coordenado pela bióloga Diana Bahia sequenciou o material genético do protozoário Leishmania amazonensis – espécie encontrada predominantemente na Amazônia que provoca uma forma mais rara e deformante de leishmaniose cutânea – e comparou com o de uma espécie-irmã, exclusiva da América Central, também marcada pelo desenvolvimento de lesões na pele semelhantes às da hanseníase. Um dos objetivos era identificar os genes, e consequentemente as proteínas por eles codificadas, que permitiriam ao parasita viver camuflado no interior das células de defesa, sem prejudicar o hospedeiro.
Usando ferramentas de bioinformática, os pesquisadores chegaram a duas proteínas candidatas a explicar por que o parasita consegue conviver harmoniosamente com as células que deveriam matá-lo. Essas proteínas pertencem à classe das chamadas heat-shock proteins, que também são produzidas pelo hospedeiro do parasita – em geral, roedores e seres humanos. Em colaboração com grupos do Laboratório Nacional de Biociências (LNBio) e do Laboratório de Genômica e Expressão da Universidade Estadual de Campinas, a equipe obteve indícios de que a proteína fabricada e liberada pelo protozoário parece imitar a do hospedeiro. Os pesquisadores suspeitam que essa imitação permitiria a ligação dessas proteínas a componentes do arsenal imunológico do indivíduo infectado, bloqueando sua ativação e silenciando a resposta inflamatória.
O estudo também investigou que genes estariam relacionados à formação de uma bolsa que abriga o parasita no interior dos macrófagos, as células de defesa que os englobam e tentam destruí-los. Nela, o parasita se multiplica e resiste a eventuais ataques do sistema imunológico. No caso da L. amazonensis e da sua espécie-irmã, a L. mexicana, essa bolsa é bem mais espaçosa do que as formadas por outras espécies de protozoários causadores da leishmaniose. “Essa pode ser uma das maneiras de o parasita subverter e enganar o sistema imunológico do hospedeiro”, conta Fernando Real, pesquisador da Unifesp e um dos autores principais do estudo que descreveu esses resultados na edição de julho da revista DNA Research.
De acordo com os pesquisadores, juntas, essas estratégias também permitiriam ao parasita viver camuflado no organismo infectado, sem desencadear os sintomas que se manifestam em pessoas com o sistema imunológico fragilizado. “Essas duas espécies deLeishmania desenvolveram estratégia para viver silenciosamente em hospedeiros vertebrados”, conta Diana. Para ela, essa convivência, pacífica especialmente para o parasita, é resultado de um longo processo evolutivo em que a morte do hospedeiro não seria vantajosa para o protozoário. “A investigação dos fatores relacionados a essa interação durável pode ser útil para entendermos como o parasita se estabelece no ser humano”, diz.
As suspeitas dos pesquisadores ainda precisam ser comprovadas experimentalmente. Um dos próximos passos agora é isolar as bolsas (vacúolos) formadas por essa espécie de Leishmania e tentar mapear seus componentes. Esse mapeamento dependia das informações do genoma da L. amazonensis, agora sequenciado e disponibilizado para a comunidade científica. “O genoma abre mais portas do que fecha”, afirma Diana. “Temos mais perguntas para investigar”.
O estudo foi financiado pela FAPESP e pelo Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq).
Projetos
1. Identificação e caracterização molecular de proteínas quinases de Trypanosoma cruzi para o estudo da comunicação celular, modelagem molecular e desenho de drogas inibidoras: estudo dos parceiros das vias de sinalização focado na invasão de EA (2007/50551-2); Modalidade Auxílio à Pesquisa – Apoio a Jovens Pesquisadores;Coord. Diana Bahia/Unifesp; Investimento R$ 532.652,38 (FAPESP).
2. Estudo dos mecanismos de fusão entre vacúolos parasitóforos heterotípicos de Leishmania spp. (2010/19335-4); Modalidade Bolsa de pós-doutorado; Coord.Renato Arruda Mortara/Unifesp; Investimento R$ 233.453,22 (FAPESP).
Artigo científico
REAL, F. et al. The genome sequence of Leishmania (Leishmania) amazonensis: functional annotation and extended analysis of gene models. DNA Research. v. 20, p. 1-15. jul. 2013.
Fonte: Revista Pesquisa - FAPESP
Registros do herbário estão disponíveis em banco de dados que reúne maiores coleções do mundo
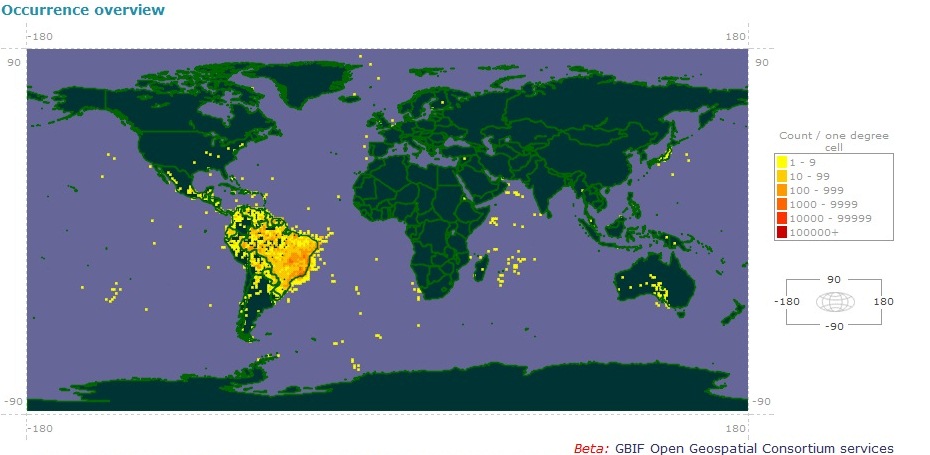
Mais do que um deslumbrante parque fundado no século XIX, o Jardim Botânico do Rio de Janeiro (JBRJ) agora está alojado junto aos principais museus internacionais. É a primeira coleção biológica brasileira a compartilhar os dados de seu herbário na Global Biodiversity Information Database (GBIF), um portal que agrega alguns dos maiores museus zoológicos e botânicos do mundo, como o Museu de História Natural de Nova York, nos Estados Unidos, e o Jardim Botânico Real de Kew, na Inglaterra. “Não é apenas o esforço de uma instituição, é um movimento em que o Brasil se engajou”, conta o biólogo Eduardo Dalcin, Coordenador do Núcleo de Computação Científica e Geoprocessamento do Instituto de Pesquisas Jardim Botânico do Rio de Janeiro.
É um movimento importante ao qual os pesquisadores brasileiros tiveram alguma resistência nos últimos anos. “Ainda é polêmico”, conta Dalcin, “tornar públicos dados sobre espécies nacionais, ameaçadas de extinção ou com interesse econômico”. Vencendo a resistência e abraçando o que ele caracteriza como “uma nova visão da ciência de compartilhamento de dados”, o JBRJ não deixa de tomar cuidado com as espécies de interesse comercial. Estas não têm latitude e longitude inseridas no banco de dados e não aparecem no mapa do GBIF. Mas outros dados de mais de meio milhão de registros estão lá e podem ser encontrados por meio de busca. “Não vamos dar indicações precisas de onde há florestas desenvolvidas de mogno”, exemplifica o biólogo.
De maneira geral, a grande importância de um banco de dados coletivo é a possibilidade de busca integrada. Ao procurar, por exemplo, registros de Caesalpinia echinata (o pau-brasil), o banco informa conter 385 registros, a maior parte na mata atlântica. Desses espécimes, 35 estão no Rio de Janeiro e 34 em Kew. Além da localização no mapa, é possível saber em que data a amostra foi coletada, onde, por quem, e o número de registro na coleção. Essa possibilidade de reunir seus dados ao maior número possível de instituições levou o JBRJ a dar o passo para o GBIF, mas não foi a primeira iniciativa coletiva do herbário carioca. “Desde 2004 estávamos noSpeciesLink do Centro de Referência em Informação Ambiental (Cria)”, conta Dalcin, “que reúne várias coleções brasileiras”.
Esse ponto de partida permitiu que o JBRJ agisse depressa assim que o Brasil firmou o acordo multigovernamental de adesão ao GBIF, no ano passado. A iniciativa nacional está sendo gerida pelo Sistema de Informação sobre a Biodiversidade Brasileira (SiBBr), um projeto do Ministério de Ciência, Tecnologia e Inovação. Do ponto de vista computacional, o nó brasileiro do GBIF estará alojado no Laboratório Nacional de Computação Científica, o LNCC, que está fazendo os ajustes necessários para lançar o portal nacional que deverá reunir outras coleções.
Dalcin considera que o acesso público aos dados é não só uma obrigação (“os dados sob a guarda da instituição precisam ser devolvidos à sociedade”), mas também um recurso valiosíssimo para estudos de conservação. Integrado a esse esforço, ele também cita esforços coletivos como a lista da flora brasileira, elaborada nos últimos anos com participação de cerca de 400 botânicos, e o catálogo da fauna que começa a ser reunido agora. “Em alguns anos pretendemos ter disponível o catálogo da vida do Brasil.” Um projeto ambicioso que dependerá do trabalho conjunto de botânicos, zoólogos e especialistas em gerenciamento de informação, entre outros.
Fonte: Revista Pesquisa - FAPESP
