 Por Fernanda Barreiro Brito Aluna do curso de graduação em Ciências Biológicas Microbiologia e imunologia, 7º período. Matéria escrita na disciplina de extensão de integração acadêmica do Curso de Bacharelado em Ciências Biológicas: Projeto de Extensão Ciência com Microbios. Creditação da extensão no Instituto de Microbiologia.
Por Fernanda Barreiro Brito Aluna do curso de graduação em Ciências Biológicas Microbiologia e imunologia, 7º período. Matéria escrita na disciplina de extensão de integração acadêmica do Curso de Bacharelado em Ciências Biológicas: Projeto de Extensão Ciência com Microbios. Creditação da extensão no Instituto de Microbiologia.
No ambiente aquático, diversas bactérias vivem em extrema hostilidade pela enorme demanda de bacteriófagos (vírus capazes de infectar esses microrganismos). Sendo assim, há uma grande pressão para que as bactérias presentes intensifiquem seus mecanismos de resistência. Dessa forma, o sistema denominado CRISPR-Cas foi observado cuidadosamente nesse ambiente e ampliou o estudo sobre a capacidade de diversos organismos procariotos, incluindo algumas arqueas, conseguirem desenvolver esse método específico de imunidade. Além disso, abriu portas para o desenvolvimento de novas aplicações no ramo da biotecnologia, principalmente no que diz respeito à capacidade de promover mecanismos de recombinação homóloga de maneira sítio-dirigida no DNA alvo.
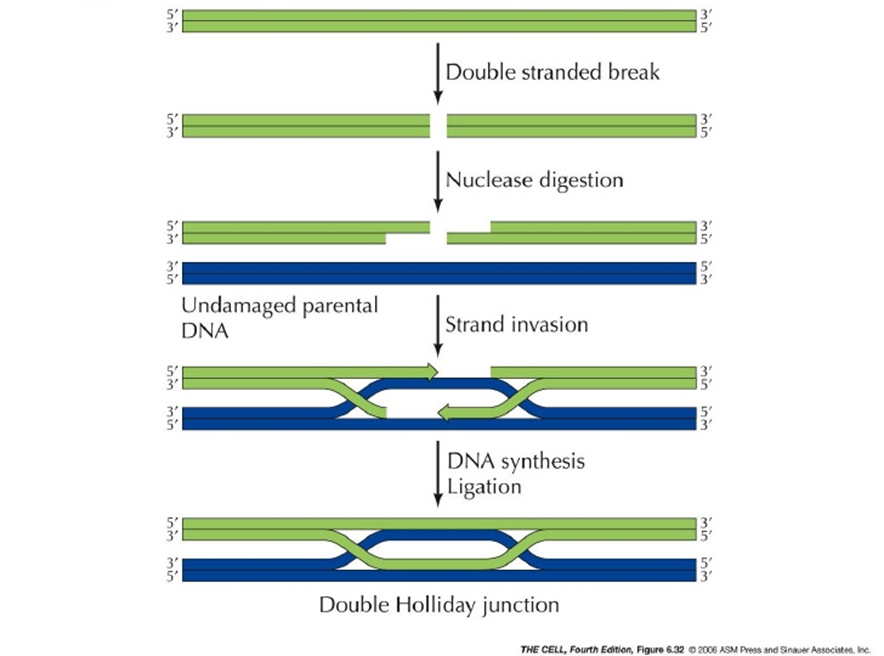
Figura 1: Reparo de DNA por recombinação homóloga.
Esse sistema é composto pela porção CRISPR, formado por repetições diretas e palindrômicas (capazes de formar grampos durante a transcrição) e espaçadores de origem exógena. Além disso, para que seja funcional, possui associação com as proteínas Cas. Em uma espécie bacteriana, as repetições são idênticas, enquanto os espaçadores são distintos.
O processo se inicia após a entrada de um DNA exógeno/invasor na célula procariota. Esse ácido nucléico é reconhecido por proteínas de reparo e posteriormente processado, o que promove o seu reconhecimento pelo complexo protéico Cas1-Cas2. Logo após, ocorre a incorporação dessa sequência exógena ao locus cromossômico onde se localiza o CRISPR, que é transcrito e passa a ter esse DNA invasor como uma região espaçadora. Durante o processo de transcrição do CRISPR, ocorre a formação de grampos, por conta das regiões palindrômicas, que são reconhecidos/processados pelas enzimas Cas 6 (no caso do CRISPR tipo I) ou pelas enzimas RNAse tipo III (no caso do CRISPR tipo II). O produto desse processamento é o crRNA proveniente dos espaçadores, sendo um ligante do patógeno que antes havia liberado seu DNA dentro da célula. Caso esse invasor infecte novamente, haverá a ligação de seu material genético com o crRNA através do reconhecimento de uma sequência específica de 2-5 nucleotídeos denominada PAM, que está presente de forma upstream ou downstream ao espaçador. Em seguida, acontece a clivagem deste pela proteína Cas 9 associada ao sistema CRISPR.
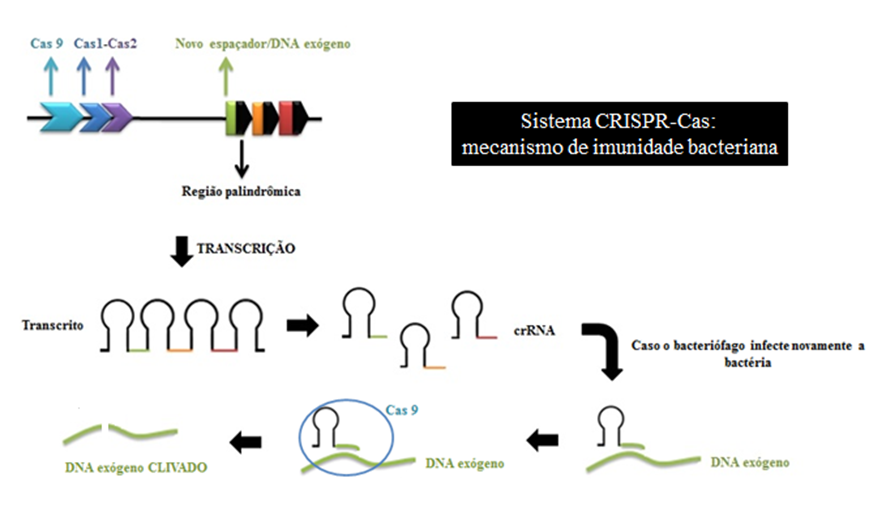
Figura 2: Mecanismo de imunidade bacteriana pelo sistema CRISPR-Cas.
Diversos estudos recentes abordam os mecanismos desse sistema, incluindo atualizações sobre os métodos iniciais de processamento do material genético exógeno e integração dos espaçadores. Entretanto, grande parte do avanço nas pesquisas sobre esse processo de imunidade de procariotos tem como objetivo a sua utilização para fins biotecnológicos. Atualmente vem sendo usado para diversas aplicações, como é o caso da construção de vetores que são capazes de expressar esse sistema CRISPR-Cas dentro de células transformadas, promovendo a clivagem de genes alvos. Esses cortes atuam no mecanismo de edição gênica sítio-dirigida, onde se inicia um processo de mutação por recombinação homóloga na região clivada. Sendo assim, o mesmo vetor que expressa o CRISPR-Cas pode expressar uma sequência nucleotídica de interesse para que seja inserida durante a mutação. Antes, métodos como o uso de Endonucleases de Dedo de Zinco (ZFN) e Nucleases com Efetores do Tipo Ativador Transcricional (TALEN) eram as escolhas para induzir esse tipo de mutagênese. Porém, o sistema CRISPR-Cas gerou novas perspectivas e promete render muitos outros avanços nesse ramo.

















